Artículos
Importancia de la genética molecular para el control del virus de Gumboro
22 August 2016Las cepas clásicas de la enfermedad infecciosa de la bursa o enfermedad de Gumboro pueden causar dos cuadros clínicos muy diferentes. Las cepas variantes provocan cuadros sub-clínicos de inmunodepresión, afectando negativamente los parámetros zootécnicos.

Artículo de Dr. Diego Hernández, Martín Hernández, Ana Marandino, Gonzalo Tomás, Ruben Pérez y Dr. Juan José Benedetti, Cátedra de Biología Molecular de la Facultad de Ciencias de la Universidad de la República Oriental del Uruguay.
La enfermedad infecciosa de la bursa o enfermedad de Gumboro es una patología viral aguda altamente contagiosa que afecta extensamente la producción de aves de postura y carne (Lukert & Saif, 2003). El agente etiológico, IBDV, es un virus del género Avibirnavirus de la familia Birnaviridae que se replica esencialmente en tejido linfoide, especialmente en la bursa de Fabricius. Este órgano es el principal centro linfopoyético, desde el cual se van a sembrar los linfocitos B en otros órganos.
Es funcional durante los primeros 6 meses de vida del ave. Luego de ese tiempo la bursa se atrofia y deja de cumplir su función. Por este motivo el virus afecta solamente aves con la bursa de Fabricius presente, replicándose y destruyendo especialmente los linfocitos B inmaduros que se desarrollan en este órgano. Así, se desarrollan cuadros de inmunodepresión, y en casos más graves, inmunosupresión (Hirai et al., 1981).
El IBDV presenta un genoma de ARN de doble cadena bisegmentado. El segmento genómico A codifica la proteína de liberación celular VP5 en un marco de lectura abierto conocido como ORFA-2 (Mundt et al., 1995) que se encuentra mayormente solapado y desfasado en un nucleótido con el ORFA-1.
Este segundo marco de lectura da origen a una poliproteína que luego de procesada genera las proteínas estructurales VP2 y VP3, y la proteasa viral VP4 (Spies et al., 1989). El segmento B codifica la RNA polimerasa viral RNA dependiente (VP1) (Morgan et al., 1988).
El serotipo patógeno de IBDV (serotipo 1) incluye tres cepas que se denominan clásicas (c), variantes (va), e hipervirulentas (vv). Ellas difieren en niveles de patogenia, y en composición genética y antigénica. Las cepas clásicas pueden causar dos cuadros clínicos bien diferentes.
Si la infección con cepas clásicas se produce en los primeros días de la vida de un ave sin anticuerpos maternales, se produce un cuadro de inmunodepresión cuya severidad es inversamente proporcional a la edad del ave. Si la infección encuentra una bursa más desarrollada, el cuadro es el que clásicamente se conoce como Gumboro clásico.
Las cepas variantes provocan cuadros sub-clínicos de inmunodepresión, afectando negativamente los parámetros zootécnicos y facilitando la acción de patógenos oportunistas que desarrollan un cuadro sobreagregado. Generalmente es difícil establecer su presencia por medio de diagnóstico clínico, provocando pérdidas un ciclo tras otro hasta no realizar un diagnóstico de laboratorio definitivo. Ambas cepas son fácilmente diferenciables antigénica y genéticamente.
Por otro lado, los vvIBDV causan graves cuadros clínicos, con alta morbilidad y mortalidad en lotes de aves jóvenes. Desde su emergencia en 1987 son motivo de gran preocupación para la industria, causado pérdidas millonarias a en todo el mundo (van den Berg et al. 2000). A pesar de desarrollar una sintomatología mucho más violenta, los vvIBDV son antigénicamente indiferenciables de los cIBDV, aunque sí presentan notorias diferencias genéticas.
Genética aplicada al diagnóstico e investigación del virus de Gumboro
El virus de Gumboro al igual que otros virus con genoma de ARN tiene una gran variabilidad genética generada principalmente por la ocurrencia de mutaciones puntuales, aunque también se han reportado casos de virus recombinantes y con reordenamientos genómicos.
Conocer las variantes que circulan mundialmente es muy útil e importante desde un punto de vista epidemiológico y de control de la enfermedad. Guiados por la hipótesis “El virus de Gumboro es prevalente en la producción avícola mundial y existen diversas cepas circulando que pueden ser caracterizadas adecuadamente desde el punto de vista genético, siendo su estudio importante para la comprensión de la variabilidad genética y evolución viral”, desde el año 2004 en el Grupo de Investigación en Genética de Microorganismos de la Facultad de Ciencias de Uruguay (http://igm.geneticafcien.com/) se ha dedicado al diagnóstico e investigación epidemiológica del virus de Gumboro.
Mediante el análisis de muestras locales y provenientes de diversos países hemos podido investigar la epidemiología molecular de Gumboro y descifrar algunos de sus mecanismos evolutivos subyacentes. Con la ayuda de técnicas de diagnóstico y caracterización molecular se desarrollan planes de control específicos para controlar eficientemente los brotes de campo, e incluso graves casos de cepas hipervirulentas (Hernández et al., 2006).
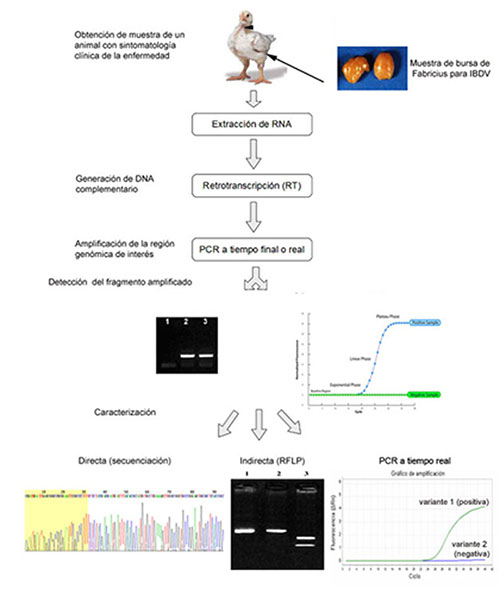
Realizando estudios comparativos de secuenciación nucleotídica y aminoacídica se pudieron identificar marcadores fuertemente asociados a las cepas hipervirulentas que han servido como referencia para la discriminación de cepas de alta y baja patogenia y para el desarrollo de técnicas de diagnóstico molecular para PCR en Tiempo Final (Figura 1) (Hernández et al., 2011) y Tiempo Real (Hernández et al., 2012; Tomás et al., 2012).
Las técnicas de detección y caracterización rápida permiten tener una respuesta para que el profesional de campo logre establecer un plan de control específicamente diseñado durante el transcurso infeccioso y disminuir el impacto del virus en crianzas posteriores.
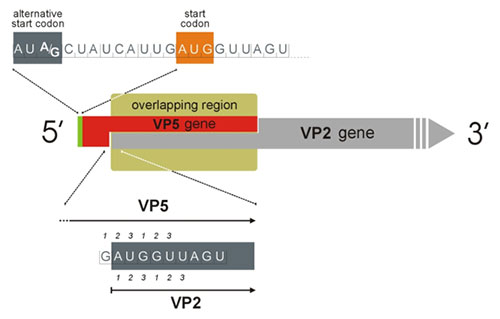
Los estudios moleculares han revelado también que algunas cepas hipervirulentas presentan cambios en el gen que codifica para la proteína de liberación celular VP5 (Figura 2) (Hernández et al., 2010). Se trata de una proteína de especial importancia por intervenir en la regulación del ciclo replicativo del virus. El aumento de virulencia de las cepas hipervirulentas es una interrogante que se mantiene sin resolver hasta nuestros días. Por la intervención de VP5 en la replicación viral, y posiblemente en la virulencia, obtener información de este gen es muy importante para entender el comportamiento del virus.
Con la aplicación de técnicas moleculares se pudo constatar que el gen vp5 se encuentra evolucionando bajo un complejo sistema de presiones selectivas, producto de su superposición con el ORF-A1 que codifica para las proteínas estructurales del virus. Más aún, este gen se habría originado posteriormente en el tiempo respecto al resto de los genes virales, utilizando un mecanismo de sobreescritura genética u overprinting.
Estos resultados colaboran en el conocimiento básico del virus, de los probables mecanismos de evolución genética, y de la dinámica del genoma de IBDV y su historia evolutiva.
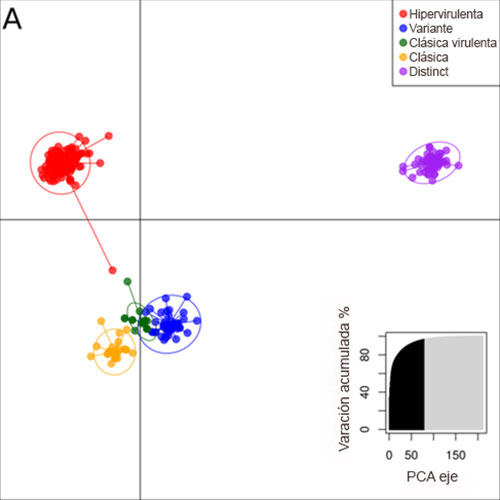
Con el uso de técnicas moleculares también se han descrito variantes y aislamientos que difieren de las cepas tradicionales. Particularmente, en los últimos años se ha observado una alta prevalencia de una cepa viral distinta a las cepas clásicas, variantes e hipervirulentas reportadas hasta ahora. Estas cepas, que fueron denominadas “distinct” (dIBDV) (Hernández et al., 2015), constituyen un linaje evolutivo independiente disperso en países de Sud, Norte y Centroamérica, Europa y Asia.
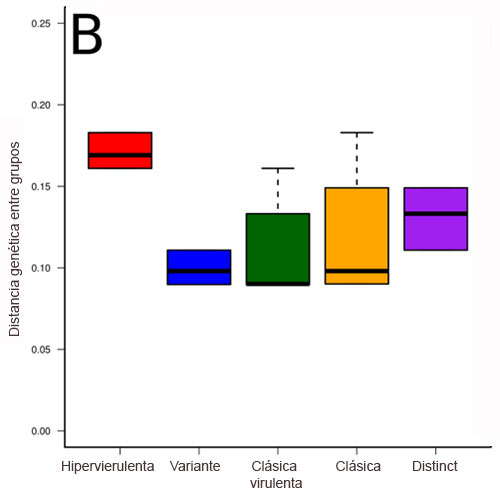
La secuenciación total del genoma (Tomás et al., 2015) ha permitido determinar que los dIBDV presentan características genómicas conservadas, que los diferencian del resto de las cepas (ej. marcadores de VP2: 272T, 289P, 290I, 296F). Análisis discriminantes de las características genéticas de este linaje certificaron que los dIBDV conforman un cluster de virus de Gumboro evolutivamente relacionados entre sí, pero separados del resto de las cepas conocidas (Figura 3).
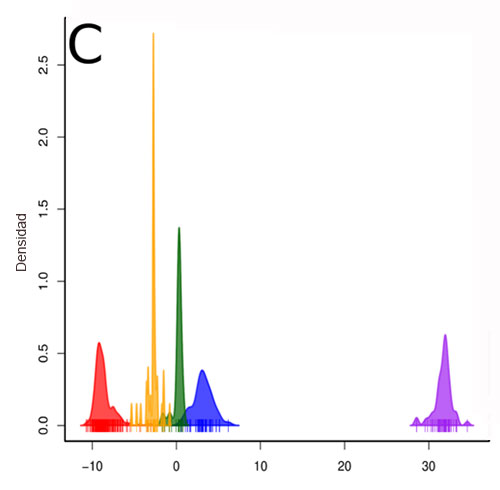
Desde un punto de vista patogénico, esta cepa tiene una manifestación subclínica, lo que complica su diagnóstico de campo y eventualmente su diferenciación del resto de las cepas subclínicas. No obstante, como ya se mencionó son fácilmente diferenciables genéticamente de manera que el diagnóstico molecular es muy conveniente para resolver el caso.
Ya se han desarrollado técnicas moleculares de diagnóstico y caracterización rápidas que establecen con un alto grado de sensibilidad la presencia de estas cepas en una muestra de campo (Tomás et al., en preparación). Finalmente, es importante tener en cuenta que dada la cantidad de cambios genéticos y aminoacídicos en la proteína VP2, donde se encuentran los principales determinantes antigénicos del virus, es posible que la cepa “distinct” sea una variante de tipo antigénico.
De confirmarse esto, se deberán revisar las vacunas disponibles para ver su nivel de control, o eventualmente evaluar el desarrollo de biológicos de control específico que cubran las características antigénicas de estas cepas. Para determinar esto se necesitan estudios posteriores que analicen el grado de relación antigénica existente entre esta cepa, las vacunas existentes y el resto de las cepas.
Desarrollar productos tecnológicos aplicables en la industria, utilizarlos para conocer la situación sanitaria y poder diseñar planes de control específicos, y avanzar en el conocimiento básico del virus de Gumboro, ha tenido y tendrá efectos positivos para el desarrollo de la industria avícola, y principalmente para una mayor comprensión de la epidemiología de este virus a nivel global.
Bibliografía
Hernández, M., A. Banda, D. Hernández, F. Panzera, Pérez R. Detection of very virulent strains of infectious bursal disease virus (vvIBDV) in commercial broilers from Uruguay. Avian Dis. 50:624-31. 2006.
Hernández, M., P. Villegas, D. Hernández, A. Banda, L. Maya, V. Romero, G. Tomás, Pérez R. Sequence variability and evolution of the terminal overlapping vp5 gene of the infectious bursal disease virus. Virus Genes 41:59-66. 2010
Hernández, M., G. Tomás, D. Hernández, P. Villegas, A. Banda, L. Maya, Y. Panzera, Pérez R. Novel Multiplex RT-PCR/RFLP Diagnostic Test to Differentiate Low- from High-Pathogenic strains and to detect Reassortant Infectious Bursal Disease Virus. Avian Dis. 55:368-74. 2011.
Hernández, M., G. Tomás, A. Marandino, Y. Panzera, D. Hernández, P. Villegas, A. Banda, Pérez R. Desarrollo de tecnología en sanidad aviar para la detección y caracterización de los virus de la Bronquitis Infecciosa y de Gumboro. Serie FPTA (http://www.inia.uy). 2012.
Hernández, M., G. Tomás, A. Marandino, G. Iraola, L. Maya, N. Mattion, D. Hernández, P. Villegas, A. Banda, Y. Panzera, Pérez R. Genetic characterization of South American infectious bursal diseases virus reveals the existence of a distinct worldwide-spread genetic lineage. Avian Path. 44(3):212-21. 2015
Hirai, M.K., T. Funakoshi, T. Nakai, & S. Shimakura. Sequential changes in the number of surface immunoglobulin-bearing B lymphocytes in infectious bursal disease virus-infected chickens. Avian Dis. 25:484-496. 1981.
Lukert, P. D., & Y. M. Saif. Infectious bursal disease. In: Diseases of poultry, 11th ed. Y. M. Saif, J. R. Glisson, A. M. Fadly, L. R. McDougald, and D. E. Swayne, eds. Iowa State University Press, Ames, IA. Pp. 161 – 179. 2003.
Morgan, M. M., I. G. Macreadie, V. R. Harley, P. J. Hudson & A. A. Azad. Sequence of the small double-stranded RNA genomic segment of infectious bursal disease virus and its deduced 90-kDa product. Virology. 163(1):240-2. 1988.
Mundt, E., J. Beyer, & H. Müller. Identification of a novel viral protein in infectious bursal disease virus-infected cells. J. Gen. Virol. 76: 437-443. 1995.
Spies, U., H. Müller & H. Becht. Nucleotide sequence of infectious bursal disease virus genome segment A delineates two major open reading frames. Nucleic Acids Res. 17(19):7982. 1989.
Tomás, G., M. Hernández, A. Marandino, Y. Panzera, L. Maya, D. Hernández, A. Pereda, A. Banda, P. Villegas, S. Aguirre, R. Perez. Development and validation of a TaqMan-MGB real-time RT-PCR assay for simultaneous detection and characterization of infectious bursal disease virus. J. Vir. Meth. 185:101-107. 2012
Tomás, G., M. Hernández, A. Marandino, D. Hernández, C. Techera, S. Grecco, Y. Panzera, R. Perez. Genome sequence of a Distinct Infectious Bursal Disease Virus. Genome Announc. 3(5), 2015.
Tomás, G., M. Hernández, A. Marandino, D. Hernández, C. Techera, S. Grecco, Y. Panzera, R. Perez. DEvelopement of a RT-qPCR assay for the specific detection of a distinct genetic lineage of the infectious bursal disease virus. En prensa.
Van den Berg, T.. Acute Infectious Bursal Disease in poultry: a review. Avian Path. 29: 175-194. 2000.
Vera, F., M. Craig, V. Olivera, F. Rojas, G. Konig, A. Pereda, A. Vagnozzi. Molecular characterization of infecious bursal disease virus (IBDV) isolated in Argentina indicates a regional lineage. Arch. Virol. version online http://dx.doi.org/10.1007/s00705-015-2449-4 2015. 2015
También le interesaría
Aspectos prácticos en el control de la enfermedad de Gumboro
Agosto 2016

 © 2000 - 2021. Global Ag MediaNinguna parte de este sitio puede ser reproducida sin previa autorización.
© 2000 - 2021. Global Ag MediaNinguna parte de este sitio puede ser reproducida sin previa autorización.